
搜索网站、位置和人员

电话: +86-(0)571-86886861 公共事务部
肾透明细胞癌(clear cell renal carcinoma, ccRCC)是肾细胞癌中最常见和最具侵袭性的亚型1。当前多种临床治疗策略,如靶向治疗和免疫治疗,仍然还无法很好地应对高度异质性的肿瘤微环境,所以在临床上并没有取得令人满意的治疗效果2,3。深入刻画ccRCC微环境中的细胞组成和基因组调控特征对于我们理解肿瘤致病机理和开发新的治疗策略具有重要意义。
近年来,单细胞测序技术飞速发展,已经能够检测出单个细胞的基因转录表达和表观基因组调控等多种维度的信息4。这种单细胞水平的多维度信息能够以更高分辨率和更广的视角来帮助解析组织发育和疾病发生发展的关键因素。最近,多个研究团队针对健康的肾脏组织,联合single-nucleus RNA-seq和single-nucleus ATAC-seq技术刻画了在肾脏发育过程基因表达和表观基因组调控的特征,并识别了驱动肾小管细胞分化和近端小管上皮细胞亚群转换的关键调控因子5,6。然而,ccRCC的单细胞多维度视角目前还没有被打开。
西湖大学生命科学学院谢琦研究员和浙江大学医学院附属第二医院泌尿外科文甲明和王博涵课题组合作作为共通讯作者(谢琦课题组龙志林为本文第一作者)近日在Cell Discovery上发表题为“Single-cell multiomics analysis reveals regulatory programs in clear cell renal cell carcinoma”的研究论文,对ccRCC在单细胞水平进行了多维度的探索。
这项研究整合了single-cell RNA-seq(scRNA-seq)和single-cell ATAC-seq (scATAC-seq)数据,描绘了ccRCC的转录组和表观基因组图谱,识别了在肿瘤细胞和多种免疫细胞中发挥重要作用的调控因子,进一步阐明了ccRCC的异质性并发现了潜在的治疗靶点。
该研究对ccRCC患者的肿瘤组织进行了配对的scRNA-seq和scATAC-seq测序,分别获得了38,097和21,272个单细胞。通过单细胞数据分析,识别了多种淋巴细胞和髓系细胞亚群。联合ccRCC标志基因CA9和拷贝数变异特征,在两种数据类型中各鉴定出了一个肿瘤细胞群体。总的来说,免疫细胞是ccRCC微环境中主要的细胞群体(>70%),肿瘤细胞只占不到10%。
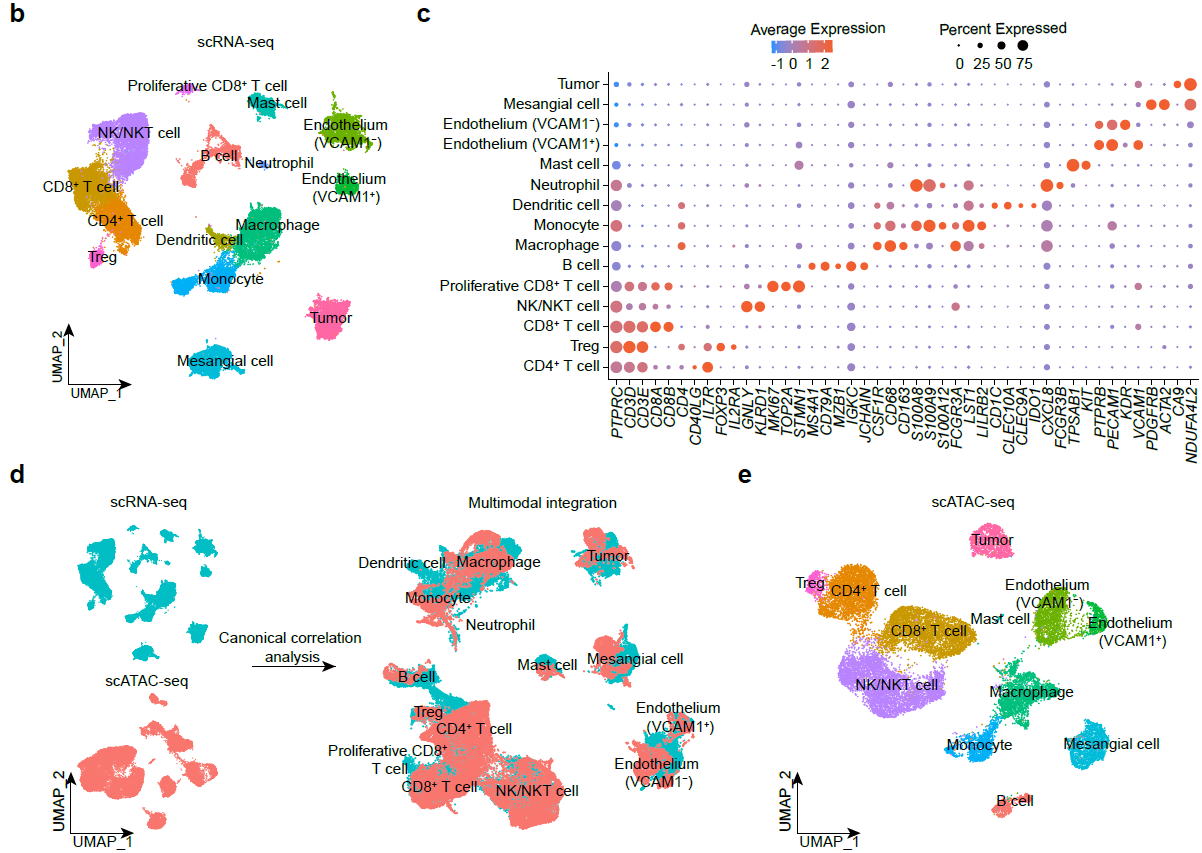
图1:ccRCC的单细胞转录和染色质可及性图谱
通过设计严格的筛选策略,并结合TCGA-KIRC生存数据,研究人员识别了4个肿瘤细胞特异富集且显著和病人更差预后生存相关的转录因子(HOXC5,VENTX,ISL1和OTP)。基于scATAC-seq捕获到的染色质开放信息,研究人员进一步识别出了这些转录因子的下游靶基因并构建了其表观调控网络。分析发现,这些转录因子的靶基因显著富集于低氧和细胞增殖信号通路,且包含多个在肿瘤细胞群体中显著高表达的基因(FXYD2和CRYAB)。此外,结合先前的scRNA-seq数据7-9,研究人员发现这些靶基因特异性地富集于肿瘤细胞群体。
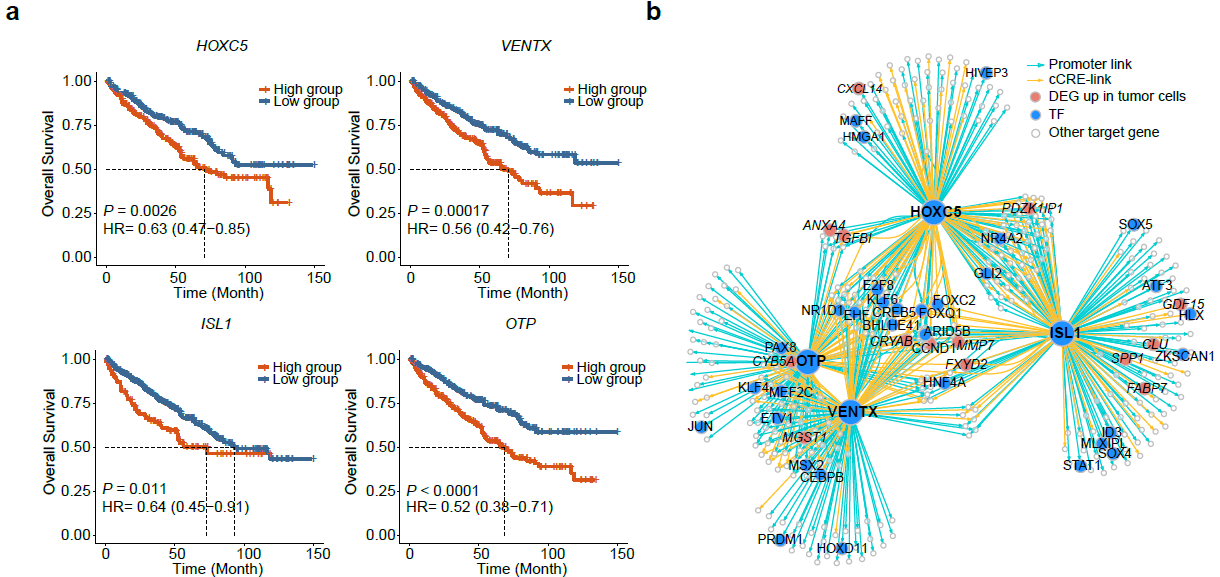
图2:4个肿瘤细胞特异富集的转录因子
为了进一步调查这些调控因子对肿瘤细胞的影响,研究人员分别在肾细胞癌细胞系和异种移植小鼠肿瘤模型中分别敲低这几个转录因子,发现能够显著降低肿瘤细胞的生长。同时结合小分子化合物扰动数据库-LINCS10,研究人员筛选出了2个已被美国食品药品监督管理局(FDA)批准用于疾病治疗的药物(homoharringtonine和mitotane)能够明显降低HOXC5,ISL1和VENTX的表达,并在两个肾细胞癌细胞系进行了进一步的验证。

图3:靶向4个转录因子抑制肿瘤生长。
此外,研究人员也刻画了对各个免疫细胞亚群的转录程序和表观特征,并在肿瘤细胞和免疫细胞亚群间识别出了多对具有潜在临床意义的配体-受体互作对。综上所述,该研究描绘了ccRCC的单细胞转录组和表观基因组图谱,识别了肿瘤细胞特异的基因转录和表观调控程序,为设计更有效的治疗策略提供了新的线索。
原文链接:https://www.nature.com/articles/s41421-022-00415-0
西湖大学谢琦研究员为本研究的共同通讯作者,西湖大学2020级博士龙志林为本文第一作者。该项研究得到了国家科学自然基金、西湖大学教育基金的支持。
西湖大学谢琦博士课题组一直从事肿瘤干细胞以及干细胞和所在的肿瘤微环境互作的相关研究,从表观遗传学,转录调控水平,翻译后修饰水平以及代谢水平对其进行了系统性研究, 取得了一系列的研究成果,研究成果以第一作者或通讯作者身份发表在Cell,Cancer Discovery, Immunity, Nature Neuroscience,Cell Research, Journal of Clinical Investigation等国际著名杂志上, 并多次应邀在干细胞领域顶级杂志Cell Stem Cell上发表评述和综述文章,详情可以参加实验室主页:https://xieqi.lab.westlake.edu.cn/。欢迎优秀的博士研究生和博士后人才加入我们的研究团队推动相关课题。有意者可以email谢琦博士(xieqi@westlake.edu.cn),长期有效。
参考文献:
1 Hsieh, J. J. et al. Renal cell carcinoma. Nat Rev Dis Primers 3, 17009, doi:10.1038/nrdp.2017.9 (2017).
2 Beksac, A. T. et al. Heterogeneity in renal cell carcinoma.Urol Oncol 35, 507-515, doi:10.1016/j.urolonc.2017.05.006 (2017).
3 Barata, P. C. & Rini, B. I. Treatment of renal cell carcinoma: Current status and future directions. CA Cancer J Clin 67, 507-524, doi:10.3322/caac.21411 (2017).
4 Kelsey, G., Stegle, O. & Reik, W. Single-cell epigenomics: Recording the past and predicting the future. Science 358, 69-75, doi:10.1126/science.aan6826 (2017).
5 Miao, Z. et al. Single cell regulatory landscape of the mouse kidney highlights cellular differentiation programs and disease targets. Nat Commun 12, 2277, doi:10.1038/s41467-021-22266-1 (2021).
6 Muto, Y. et al. Single cell transcriptional and chromatin accessibility profiling redefine cellular heterogeneity in the adult human kidney. Nat Commun 12, 2190, doi:10.1038/s41467-021-22368-w (2021).
7 Young, M. D. et al. Single-cell transcriptomes from human kidneys reveal the cellular identity of renal tumors. Science361, 594-599, doi:10.1126/science.aat1699 (2018).
8 Braun, D. A. et al. Progressive immune dysfunction with advancing disease stage in renal cell carcinoma. Cancer Cell39, 632-648 e638, doi:10.1016/j.ccell.2021.02.013 (2021).
9 Bi, K. et al. Tumor and immune reprogramming during immunotherapy in advanced renal cell carcinoma. Cancer Cell39, 649-661 e645, doi:10.1016/j.ccell.2021.02.015 (2021).
10 Subramanian, A. et al. A Next Generation Connectivity Map: L1000 Platform and the First 1,000,000 Profiles. Cell 171, 1437-1452 e1417, doi:10.1016/j.cell.2017.10.049 (2017).
最新资讯
大学新闻
大学新闻
大学新闻

















